Quello che facciamo è cercare corrispondenze
 Continua la chiacchierata con Pio D’Adamo.
Continua la chiacchierata con Pio D’Adamo.
A che punto siete arrivati?
Abbiamo fatto le analisi con il veracode, cioè le analisi mirate sugli SNP, in più abbiamo iniziato a fare quelle con i vetrini. Quello che dovremmo fare ora a breve è incrociare i dati. Prendiamo tutte le persone che, per esempio, hanno detto la ricotta mi piace molto (punteggio 9) e prendiamo tutte quelle che hanno detto proprio non mi piace (punteggio 1). Uno alla volta testiamo tutti gli SNP in tutte le persone. Se scopro che, per esempio, per uno specifico SNP il 30% di quelli che hanno detto 1 ha come base la T e il 70%, sempre in quello SNP specifico, ha la C e lo stesso avviene in quelli a cui piace molto, significa che questo SNP non è coinvolto per nulla perchè ho circa la stessa percentuale di basi in chi non ama la ricotta e in chi invece la adora. In pratica vuol dire che lui non influenza nulla. Se la situazione in uno dei due casi è invece l’opposto, la domanda da porsi è perchè chi, per esempio, ha T tende a preferire la ricotta e invece quelli che hanno C tendono a schifarla? Quando si trova una simmetria di questo tipo significa che probabilmente questo SNP ha a che fare con la sensazione di piacevolezza associata e allora vado a vedere dov’è questo SNP, in che gene sta, che fa questo gene, perché è legato a questa cosa. E lo inizio a studiare. La cosa diventa interessante quando gusti simili danno grafici con lo stesso SNP.
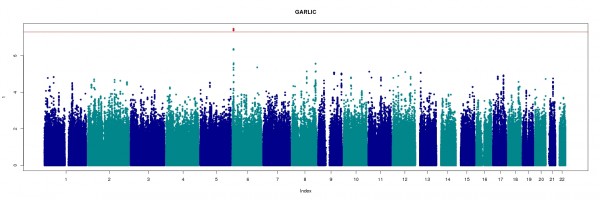 Pio ci spiega che i dati ottenuti dal processamento dei campioni vengono rappresentati tramite un manhattan plot, un grafico chiamato così perché ricorda le linee tracciate dai grattacieli sull’orizzonte. In un manhattan plot ogni colore identifica un cromosoma diverso, mentre ogni puntino uno SNP testato. Sull’asse delle ascisse è indicata la posizione fisica sul genoma; su quello delle ordinate, invece, l’intensità dell’associazione tra quello SNP e ciò che si sta studiando. Più è forte l’associazione (chiamata anche simmetria), più lo SNP starà in alto sull’asse delle Y. Se invece non c’è simmetria e tutti gli SNP sono uguali il puntino rimarrà in basso.
Pio ci spiega che i dati ottenuti dal processamento dei campioni vengono rappresentati tramite un manhattan plot, un grafico chiamato così perché ricorda le linee tracciate dai grattacieli sull’orizzonte. In un manhattan plot ogni colore identifica un cromosoma diverso, mentre ogni puntino uno SNP testato. Sull’asse delle ascisse è indicata la posizione fisica sul genoma; su quello delle ordinate, invece, l’intensità dell’associazione tra quello SNP e ciò che si sta studiando. Più è forte l’associazione (chiamata anche simmetria), più lo SNP starà in alto sull’asse delle Y. Se invece non c’è simmetria e tutti gli SNP sono uguali il puntino rimarrà in basso.
Cosa significa se due gusti simili danno grafici con lo stesso SNP?
Prendiamo per esempio il limone e il pompelmo. Tu scopri che tendenzialmente a chi piace il limone piace anche il pompelmo. Poi vai a vedere il grafico del limone e ti esce un certo SNP, vai a vedere il grafico del pompelmo e ti esce lo stesso SNP. La cosa diventa molto interessante, perché tu già a priori sapevi che tendenzialmente a chi piace uno piace anche l’altro. E qui ti stanno dicendo che lo stesso SNP, quindi lo stesso gene, probabilmente, è responsabile di tutte e due le cose. E tu dici: eh cacchiarola ha senso!
“Pio..sarebbe bello!” aggiunge Antonietta Robino, biologa, seduta due sedie più in là. E Pio risponde “Sarebbe bello sì (scoppiano a ridere) .. ma … chi può dirlo!”
E per quanto riguarda la percezione dell’amaro?
Il PTC si sa che è sotto il controllo del TAS2R38 ma non è un controllo totale. È più del 70%. questo vuol dire che c’è un 30% che se io vado a verificare il polimorfismo di quel gene in 100 persone e so che tutti e 100 dovrebbero essere non taster, in realtà trovo 70 persone non taster e 30 che pur avendo questo SNP qua saranno invece taster. E non si sa perchè, o meglio si sa che probabilmente c’è un altro gene, non importante come questo, che però modula. Ma nessuno sa qual è.
E come si procede in questo caso?
Una delle cose è provare a trovarlo. Prendi tutti quelli che non quadrano, cioè il 30%, e provi a vedere se hanno in comune qualcosa. Se quel 30% ha in comune qualcosa da qualche altra parte, allora probabilmente è lui il responsabile.
La seconda cosa che si può fare è: dato che noi abbiamo le preferenze alimentari per tutte le nazioni e sappiamo anche se sono taster o non taster e quanti sono taster e quanti non lo sono, possiamo provare a vedere se le differenti preferenze alimentari possono essere spiegate almeno in parte proprio dalla diversa percentuale di taster e non taster in questi posti.




