Un salto nella genetica
Mentre in laboratorio si continuano a processare campioni di DNA e si inizia l’analisi dei primi dati, cerchiamo di capire meglio chiacchierando con Pio D’Adamo (responsabile del processo di genotipizzazione) come funziona e cosa avviene in questa fase della ricerca.
 Abbiamo visto che si stanno usando contemporaneamente due diversi protocolli per processare i campioni di DNA. Ma perché?
Abbiamo visto che si stanno usando contemporaneamente due diversi protocolli per processare i campioni di DNA. Ma perché?
Facciamo un passo indietro e riprendiamo alcune nozioni base di genetica.
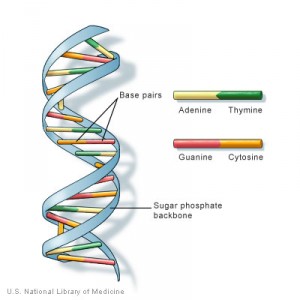
Il DNA è formato da quattro basi azotate, chiamate anche nucleotidi: adenina (A), guanina (G), citosina (C) e timina (T). La disposizione in sequenza di questi quattro “mattoncini” costituisce l’informazione genetica, il codice che fa le persone. È un po’ come avere un libro in cui ci sono scritte le istruzioni per creare un corpo umano in grado di funzionare bene.
In alcuni punti, come per esempio in quelle zone in cui sono codificati pezzi di geni, queste sequenze sono fisse e uguali per tutti gli individui. Se cambiano, infatti, si va incontro a malattie genetiche che compromettono il corretto funzionamento del corpo umano.
Ci sono altri punti, invece, detti di ipervariabilità (attualmente sono circa 7 milioni), in cui ci possono tranquillamente essere dei cambiamenti. Sono zone dentro o fuori i geni: non è infatti obbligatorio che tutte le aree del gene siano uguali, alcune possono essere fatte anche in modo alternativo. Un esempio sono i gruppi sanguigni: c’è A, c’è B, c’è 0 ma non significa che uno sia migliore di un altro.
Tra tutte le variabilità possibili a noi interessano soprattutto quelle in cui a cambiare è un’unica base e, nello specifico, dove si verifica un tipo di cambiamento chiamato SNP (single nucleotide polimorphism – polimorfismo di una singola base).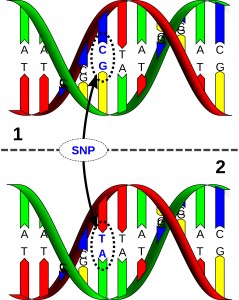
In uno SNP per esempio di tipo T/C tutta la sequenza sarà uguale tranne nel punto in cui qualcuno avrà come base una T mentre qualcun altro avrà una C.
Questi punti di polimorfismo sono molto interessanti perché se presenti all’interno dei geni possono portare alla produzione di proteine diverse. Un gene per funzionare e produrre una proteina deve essere tradotto e questo avviene leggendo la sequenza delle basi che lo compongono a blocchetti di tre (per esempio AAG – TTT). E sono proprio queste a determinare il tipo di amminoacido da attaccare. Ci sono alcune variazioni che non cambiano l’amminoacido e quindi avere una base o un’altra è indifferente. Altre, invece, lo modificano perchè l’informazione codificata è un’altra.
Nel caso della percezione dell’amaro, attraverso l’analisi di queste variazioni si potrebbe scoprire che, per esempio, tutti quelli che hanno come base T (o C) presentano anche un’attività superiore o inferiore di un determinato recettore e questo potrebbe spiegare i diversi gradi di percezione dell’amaro.
In sintesi, quindi, quello che si vuole capire attraverso queste analisi è il tipo di base presente in queste posizioni cruciali all’interno dei geni del gusto.
Il Veracode è un protocollo per verificare ipotesi. È un’analisi mirata e a bassa densità sulla base di conoscenze pregresse che possono venire dalla letteratura o da altri studi.
 Durante la fase liquida del protocollo vengono scansionati migliaia di micro cilindretti della lunghezza di 240 micron e diametro di 28 micron contenuti in un liquido. Dentro ognuno di questi cilindretti è serigrafato un codice a barre tridimensionale che lo identifica in modo univoco mentre sulla sua superficie è collocato un filamento di DNA molto specifico.Il DNA ha una struttura a doppia elica caratterizzata da un legame forte e specifico tra le due parti: un’elica singola si attaccherà solo ed esclusivamente alla sua complementare. Sfruttando questa complementarietà tra le basi, per cui una sequenza del tipo TCCCAA si attaccherà solo ad una del tipo AGGGTT, posizioniamo sui cilindretti delle eliche singole tutte uguali tra di loro e tutte specifiche per un determinato gene che, nel nostro caso, sarà un gene del gusto. A questo punto si rompe il DNA e lo si sottopone a un processo che culmina con l’attribuzione di una fluorescenza colorata verde o rossa, rispettivamente alle basi T e C.
Durante la fase liquida del protocollo vengono scansionati migliaia di micro cilindretti della lunghezza di 240 micron e diametro di 28 micron contenuti in un liquido. Dentro ognuno di questi cilindretti è serigrafato un codice a barre tridimensionale che lo identifica in modo univoco mentre sulla sua superficie è collocato un filamento di DNA molto specifico.Il DNA ha una struttura a doppia elica caratterizzata da un legame forte e specifico tra le due parti: un’elica singola si attaccherà solo ed esclusivamente alla sua complementare. Sfruttando questa complementarietà tra le basi, per cui una sequenza del tipo TCCCAA si attaccherà solo ad una del tipo AGGGTT, posizioniamo sui cilindretti delle eliche singole tutte uguali tra di loro e tutte specifiche per un determinato gene che, nel nostro caso, sarà un gene del gusto. A questo punto si rompe il DNA e lo si sottopone a un processo che culmina con l’attribuzione di una fluorescenza colorata verde o rossa, rispettivamente alle basi T e C. Immettendo ora le sequenze di DNA colorate in maniera base specifica nel liquido con i cilindretti si osserva come ogni sequenza cerchi la sua complementare. Se non la trova, si perde, se la trova, vi si attacca e, dato che ha un colore specifico per la base che cambia, leggendo il cilindretto si sa che tipo di gene stiamo osservando mentre leggendo il colore si sa che quel gene in quel punto ha una base T o una C.
Immettendo ora le sequenze di DNA colorate in maniera base specifica nel liquido con i cilindretti si osserva come ogni sequenza cerchi la sua complementare. Se non la trova, si perde, se la trova, vi si attacca e, dato che ha un colore specifico per la base che cambia, leggendo il cilindretto si sa che tipo di gene stiamo osservando mentre leggendo il colore si sa che quel gene in quel punto ha una base T o una C.
L’analisi con il Veracode presenta però una carenza. Essendo un protocollo basato su conoscenze pregresse c’è il rischio di perdere delle informazioni preziose se le variazioni responsabili di un certo comportamento non sono presenti nell’insieme dei dati che si è scelto di processare.
Con un approccio d’insieme com’è quello con i vetrini, invece, si ha un quadro completo dell’intero genoma dell’individuo da cui poi si può decidere di scendere nel dettaglio di specifiche aree con il Veracode.
Ed è per questo che vengono usati entrambi i protocolli.




